This study investigates the association between genetic polymorphisms located on chromosomes 22 and 25 and intramuscular fat (IMF) content in Hanwoo cattle. Three single nucleotide polymorphisms (SNPs), rs799031002 (ACVR2B gene), rs209494671, and rs210204777 (both in the CCL24 gene), were identified and analyzed for their correlation with marbling score (MS), longissimus dorsal IMF (D-IMF), and semimembranosus IMF (S-IMF). A total of 500 Hanwoo steers, reared under standardized conditions, were used for this analysis. Fat content measurements followed AOAC (2000) protocols, while marbling score was evaluated using a 1–9 grading system.
Linear regression models were applied to assess the genetic effect of these SNPs on the target phenotypes. Although rs799031002 showed a noticeable difference in fat content between genotypes, particularly for GG homozygotes, none of the SNPs exhibited statistically significant associations with MS, D-IMF, or S-IMF at a 0.05 significance level. However, rs209494671 and rs210204777 demonstrated borderline significance for marbling score (p ≈ 0.0598), indicating a potential trend that warrants further investigation.
The study highlights the need for expanded sample sizes and deeper genomic exploration to validate these findings. The results provide foundational insights for genomic selection strategies aimed at improving the quality and marbling characteristics of Hanwoo beef, ultimately contributing to the efficiency and precision of genetic improvement programs in Korean cattle.
최근 한국의 급격한 경제 성장과 함께 국민의 식생활 수준이 눈에 띄게 향상되면서 육류 소비 패턴에도 큰 변화가 나타나고 있다. 과거에는 소비량(quantity)에 중점을 두었던 쇠고기 소비가 이제는 품질(quality)을 중시하는 방향으로 전환되고 있다(오동협, 2014). 특히 한우고기는 소비자들 사이에서 뛰어난 육질과 풍미로 인기를 얻고 있으며, 이는 고급육 소비 트렌드와 맞물려 한우 고급육 생산에 대한 수요가 증가하고 있음을 의미한다. 한우 고급육은 도체등급 판정에서 육질 1등급 이상으로 평가되는 고품질 쇠고기로 정의되며, 이는 우리 국민이 선호하는 맛과 풍미를 객관적으로 나타내는 지표가 된다.
쇠고기의 품질은 육색, 지방색, 연도 및 근내지방 등 다양한 요소에 의해 좌우되며(Hocquette 등, 2005; Geay 등, 2001), 그 중에서도 근내지방은 쇠고기의 풍미와 연도를 결정하는 중요한 요인으로 인정받아 왔다(Tatum 등, 1982). 근내지방은 고기의 풍미와 부드러움을 향상시켜 소비자 만족도를 높이는 반면, 등지방 및 내장지방과 같은 불필요한 지방은 경제적 손실 및 생산 효율 저하를 유발한다. 이에 따라 기존에는 소의 비육기간 연장 및 고에너지 사료급여 프로그램 등을 통해 근내지방도를 개선하려는 노력이 지속되어 왔으나(Kim 등, 2005), 이들 방법은 기대한 바와 같이 근내지방은 증가시키지만 불가식 지방의 증가라는 부작용을 동반하여 전반적인 쇠고기 생산 효율성 측면에서는 한계가 있었다.
이러한 상황에서 한우 고급육 생산을 위한 새로운 접근법으로 유전적 개량 프로그램(Genetic Improvement Program)이 제시되고 있다(Park et al., 2013). 그러나 한우의 근내지방과 같은 양적형질(quantitative traits)은 가축이 도축되어야만 측정할 수 있는 형질이기 때문에, 기존의 후대검정(progeny testing) 방식은 후대 생산과 24개월 이상의 검정 기간을 필요로 하여 평균 약 5년 정도의 긴 시간이 소요된다. 이와 같이 긴 세대간격과 낮은 선발 강도는 유전적 변화 속도를 저해하며, 따라서 생산성과 경제성을 동시에 만족시키기 어렵다는 문제점을 내포하고 있다.
이러한 한계를 극복하고자 최근 육종학자들은 가축의 유전체 정보를 활용하여 개체의 생산능력 및 형질을 조기에 예측할 수 있는 마커도움선발(Marker Assisted Selection) 및 유전체도움선발(Genomic Selection) 기법을 적극 도입하고 있다. 이들 방법은 근육 내 지방 축적과 관련된 유전자들의 생리·생화학적 기능을 규명하고, 해당 유전자에서 발생하는 단일염기다형성(SNP) 등 유전변이 정보를 활용하여 형질과의 통계적 연관성을 분석하는 것을 기본 원리로 한다(Lee 등, 2006; Zhang 등, 2008). 예를 들어, 근육 내 지방 합성에 중요한 역할을 하는 FABP4, GLUT4 등 주요 유전자의 발현 패턴과 변이 정보를 통합 분석함으로써, 형질에 큰 영향을 미치는 유전적 요인을 조기에 식별하고 이를 통해 개체 선발 및 육종 프로그램의 효율성을 크게 향상시킬 수 있다.
특히, 한우 염색체 22번은 근내지방 형질과 관련된 여러 유전자들이 위치한 주요 영역으로 주목받고 있다. 염색체 22번에서 검출된 유전변이 정보는 한우의 근육 내 지방 축적에 영향을 미치는 분자적 메커니즘을 규명하는 데 중요한 단서를 제공할 것으로 기대된다. 이러한 변이들이 실제로 근내지방 형질과 어떠한 연관성을 가지는지 통계적 분석을 통해 밝힘으로써, 한우 고급육 생산을 위한 효과적인 유전적 개량 전략을 마련할 수 있을 것이다.
본 연구는 한우 염색체 22번에서 검출된 유전변이 데이터를 기반으로, 이들 변이와 한우 근육 내 지방형질 간의 통계적 연관성을 분석하는 데 중점을 두고 있다. 이를 위해, 먼저 고해상도 유전체 분석 기술을 적용하여 염색체 22번 내 변이 분포 및 밀도를 정밀하게 파악하고, 이들 유전변이와 근내지방 표현형 간의 상관관계를 통계적 모델을 통해 분석하며, 연관성이 높은 변이에 대해 추가적인 기능적 검증을 실시할 계획이다.
본 연구의 결과는 한우 개량 분야에서 근내지방 형질과 관련된 주요 유전적 요인을 명확히 밝힘으로써, 단기적으로는 마커도움선발(Marker Assisted Selection) 및 유전체도움선발(Genomic Selection) 프로그램의 정확도를 높이는 데 기여할 것으로 기대된다. 또한, 장기적으로는 한우 고급육 생산을 위한 효율적이고 경제적인 유전적 개량 전략 수립에 기초 자료로 활용될 수 있을 것이다. 나아가, 이러한 연구 접근법은 다른 가축 품종이나 육질 개선 연구에도 응용 가능성이 높아, 전반적인 축산업 발전에도 긍정적인 영향을 미칠 것으로 전망된다.
이 연구는 한우의 등심과 설도에서 측정된 고기의 근내지방과 지방함량이라는 두 가지 중요한 양적형질과 한우 염색체 22번, 25번에서 검출된 유전변이 (rs799031002, rs209494671, rs210204777)와의 연관성을 분석하였다(표1). 한우 염색체 22, 25번에서 검출된 유전변이가 한우 등심, 설도의 지방함량과 통계적 연관성이 있는지 회귀분석을 통하여 분석하고 그 효과를 검정하기 위하여 수행하였다. 따라서 본 연구의 가설은, 아래와 같다.
귀무 가설($H_0$): 한우 염색체 22, 25번 유전변이는 한우고기 지방함량과 연관성이 없다.
대립 가설($H_1$): 한우의 염색체 22, 25번 유전변이는 한우고기 지방함량에 유전적으로 영향을 미친다.
본 연구에서는 동일한 환경에서 자란 한우 100마리를 대상으로 하였으며, 이들 한우의 등심과 설도 부위에서 지방 함량을 측정하였다. Bhuiyan et al. (2018)의 연구에서도 유사하게 longissimus dorsi (등심) 근육에서 측정한 지방함량과 근내지방도를 공시재료로 사용하였다. 공시재료는 총 1,0000두 자료에서 임의적으로 500개의 표본을 추출하여 유전변이와 등심, 설도의 지방함량과의 연관성 분석을 수행하였다.
등심 및 설도의 지방함량(조지방) 측정: 근내지방도의 평가와 함께 조지방 함량도 중요한 요소로 고려됩니다. 본 연구에서는 AOAC(2000)의 표준 방법에 따라 각 위치에서 조지방 함량을 측정하였으며, 흉추 13번 부위에서 채취된 등심조직으로부터 실험적으로 조지방 함량을 측정하였습니다. 이는 고급 한우의 품질과 직결되는 중요한 변수로, 평가사의 주관적 판단을 보완하는 과학적 지표로 사용된다 (Cho et al., 2010). 근내지방도는 등급판정사의 주관적 판단으로 판정하였고, 1~9점으로 평가된다.
본 연구에서 조사된 한우 근내지방 표현형에 대한 유전자형의 효과를 분석하기 위하여 R 소프트웨어를 사용하여 아래와 같은 선형회귀모형(Linear regression model)으로 연관성 분석을 수행하였다. 유전자형에 따른 각 표현형 가의 최소제곱평균(Least square mean)과 표준오차를 추정하여 평균간 최소유의차 검정을 실시하였다.
$$Y_{ij}=\mu + bG_i + e_{ij}$$
$Y_{ij}$ : 각 표현형 관찰값
$\mu$ : 전체 평균
$G_i$ : 유전자형 효과
$b$ : 유전자형에 대한 회귀 계수
$e_{ij}$ : 임의오차, $N\sim(0,σ_e^2)$
Table 1에서 보여주는 것과 같이 본 연구에서 사용한 유전변이는 염색체 22번과 25번에서 총 세 개의 유전 변이가 검출되었다. 염색체 22번 염기서열을 기반으로 한 위치인 11849704bp에 위치한 rs799031002는 ACVR2B 유전자에 속하며, 참조 염기(REF)는 C, 변이 염기(ALT)는 G이다. 염색체 25번 염기서열을 기반으로 한 위치 34066877bp에 위치한 rs209494671과 34066884bp의 rs210204777는 모두 CCL24 유전자에 속하며, 각각 참조 염기 G와 C가 변이 염기 A와 T로 대체되었다.
Table 1. Genetic variants detected on chromosome 22, and 25 in Hanwoo
| CHROM | POS | REF | ALT | Ref SNPID | GENE | GENEID |
|---|---|---|---|---|---|---|
| 22 | 11849704 | C | G | rs799031002 | ACVR2B | ENSBTAG00000018105 |
| 25 | 34066877 | G | A | rs209494671 | CCL24 | ENSBTAG00000026275 |
| 25 | 34066884 | C | T | rs210204777 | CCL24 | ENSBTAG00000026275 |
Figure 1 각 유전자형에 대한 시각적 분포를 보여주고 있다. 그림 1의 (A) rs799031002, (B) rs209494671, (C) rs210204777의 유전변이가 한우의 근내지방도, 등심의 지방함량, 그리고 설도의 지방함량과의 연관성을 상자그림으로 표현하였다. 상자그림(Boxplot)의 결과 rs799031002 유전변이의 경우CC, CG type은 근내지방 평균에서 큰 차이를 보이지 않았으나, GG유전자형에서는 매우 큰 차이를 보였다. 이러한 결과는 등심의 지방함량, 설도의 지방함량에서도 같은 결과를 보여 주었다. 특히 등심의 지방함량에 대해서 CC, GC 유전자형 그룹은 지방함량이 약 15~17%를 보였지만, GG유전자형에서는 28%의 평균값을 보였다. 이는 GG 유전자형에서 등심 지방함량에 큰 영향을 미치는 결과라 할 수 있을 것이다 (Figure 1(A)).
한우 염색체 25번에서 검출된 rs209494671 유전변이의 경우, AG유전자형은 근내지방도의 평균이 6.7이었고, GG유전자형은 5로 두 유전자형에서 약 1.7정도의 근내지방의 차이를 보였다. 또한 등심의 지방함량의 경우, AG유전자형 그룹은 16%의 지방함량을 보였고, GG 유전자형의 경우 15.2%의 평균을 보였다. 이는 두 유전자형 간의 차이가 크지 않음을 보여주는 결과이다.
한우 염색체 25번에서 검출된 rs210204777 유전변이의 경우, CC유전자형의 경우 근내지방 평균이 4.5, 그리고 TC 유전자형의 경우 6.5의 근내지방도 평균을 보였다. 그리고 등심의 지방함량의 경우, CC 유전자형은 15.3%, 그리고 CC유전자형은 15.6%의 지방함량 평균을 보였다. 이러한 차이는 유전자형 별로 표현형의 차이가 크지 않음을 보여주는 결과이다.
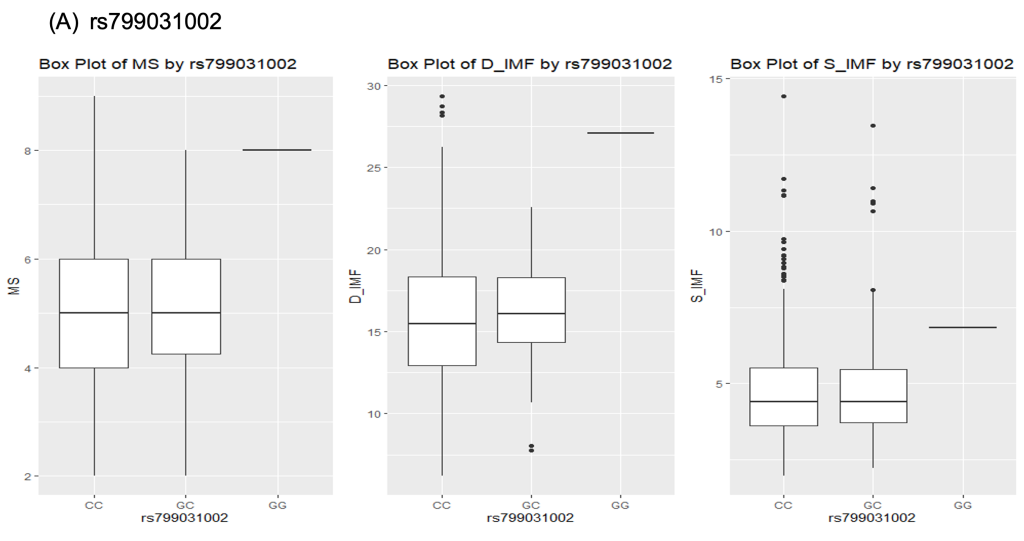
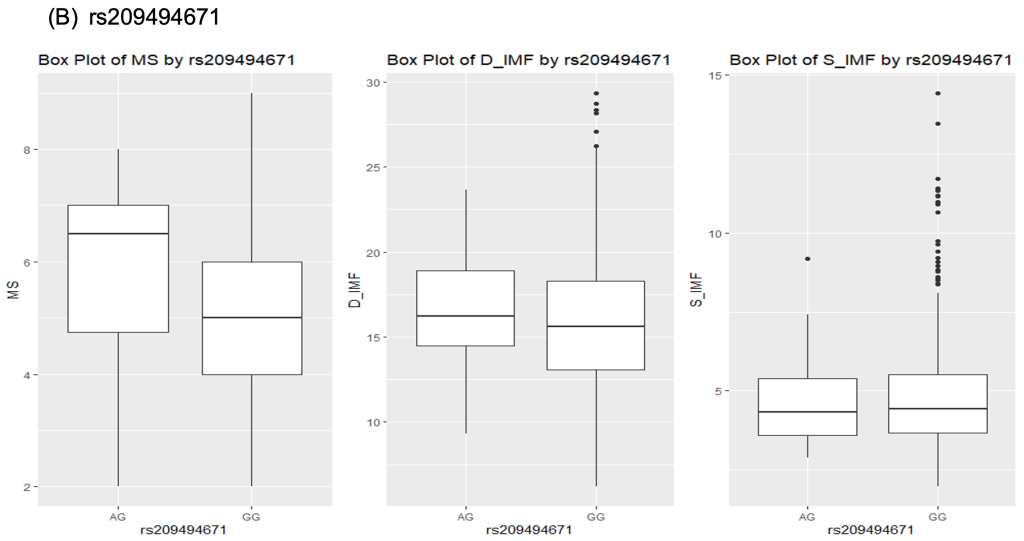
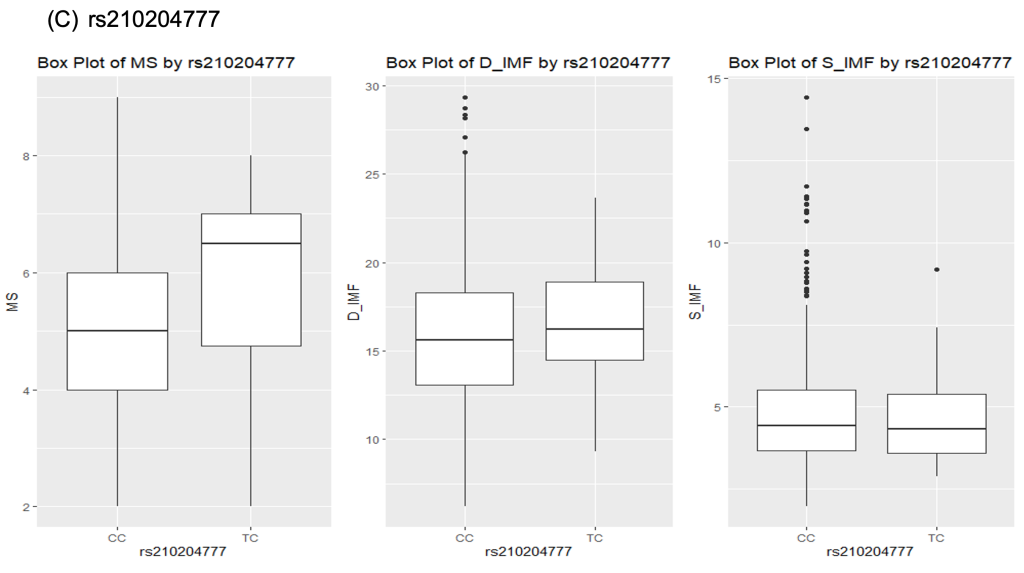
Fig. 1. Boxplot for association between genotypes and marbling score, D_IMF and S_IMF in Hanwoo
Table 2에서 보여주는 것과 같이 rs799031002의 대립유전자 빈도에 대해서, C 대립유전자는 92%, G 대립유전자는 8%의 빈도로 나타났고, 유전자형에 대해서는 CC 405(0.84)개, GC 74(0.15)개, GG 1(0.01)개로 관찰되었다. rs209494671의 경우, G 대립유전자는 98%, A 대립유전자는 2%로 매우 높은 G 대립유전자 빈도가 나타났다. 그리고 유전자형 빈도는 GG 460개, AG 20개, AA는 관찰되지 않았다. rs210204777에서도 C 대립유전자가 98%, T 대립유전자가 2%의 빈도를 보였으며, 유전자형 빈도는 CC 460개, TC 20개, TT는 관찰되지 않았다.
Table 2. Genotype and allele frequencies of three genetic variants in Hanwoo
| rs799031002 | rs209494671 | rs210204777 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Genotypes | Allele Freq | Genotypes | Allele Freq | Genotypes | Allele Freq | ||||||||||
| CC | GC | GG | C | G | GG | AG | AA | A | G | CC | TC | TT | C | T | |
| 405 | 74 | 1 | 0.92 | 0.08 | 460 | 20 | 0 | 0.02 | 0.98 | 460 | 20 | 0 | 0.98 | 0.02 | |
세 개의 유전변이에 대해서 마블링 점수(Marbling score), 등심지방함량(D-IMF), 설도지방함량(S-IMF)과의 연관성을 분석한 결과는 다음과 같다. rs799031002 유전변이는 마블링 점수에 대한 회귀계수는 0.07580, p-value는 0.673로 통계적으로 유의하지 않았다. D-IMF에 대한 회귀계수는 0.6088, 그리고 p-value = 0.228, S-IMF에 대한 회귀계수는 0.20371, 그리고 p-value = 0.367로 역시 유의하지 않았다. 그리고 한우 유전체 25번의 rs209494671 유전변이의 경우, 마블링 점수에 대한 회귀계수는 0.62609이며, p-value는 0.05로 0.05에 가까운 경향성을 보였다. D-IMF와 S-IMF에 대해서는 회귀계수 0.7132와 -0.09413으로, 각각 p-value가 0.447과 0.822로 나타나 통계적 유의성이 확인되지 않았다. 더욱이, rs210204777 유전변이의 경우, rs209494671과 동일한 결과를 보였는데, 마블링 점수에서 회귀계수는 0.62609, p-value는 0.0598로 경향성을 보였으나 유의하지 않았으며, 다른 두 지표에서도 통계적 유의성은 나타나지 않았다.
Table 3. ANOVA table for regression analysis for three genetic variants
| SNP | Marbling score | D-IMF | S-IMF | |||
|---|---|---|---|---|---|---|
| Regression coefficient | p-value | Regression coefficient | p-value | Regression coefficient | p-value | |
| rs799031002 | 0.07580 | 0.673 | 0.6088 | 0.228 | 0.20371 | 0.367 |
| rs209494671 | 0.62609 | 0.0598 | 0.7132 | 0.447 | -0.09413 | 0.822 |
| rs210204777 | 0.62609 | 0.0598 | 0.7132 | 0.447 | -0.09413 | 0.822 |
이번 분석에서 세 개의 SNP 변이는 한우의 마블링 점수와 등심, 설도 지방함량에 대해서 약간의 경향성을 보였으나, 통계적으로 유의한 결과는 보이지 않았다. 그러나 rs209494671과 rs210204777의 경우 마블링 점수에서 약간의 통계적 유의성의 경향이 관찰되어, 추가 연구를 통해 더 많은 표본을 이용하여 유전변이의 효과를 검증할 필요가 있다.
# 데이터 로드
data <- read.table(“data-snp.txt”, header = TRUE)
# 데이터 요약
summary(data)
str(data)
# Box Plot 그리기
library(ggplot2)
# rs799031002와 형질 간의 Box Plot
p1=ggplot(data, aes(x = rs799031002, y = MS)) +
geom_boxplot() + labs(title = “Box Plot of MS by rs799031002”, x = “rs799031002”, y = “MS”)
p2=ggplot(data, aes(x = rs799031002, y = D_IMF)) +
geom_boxplot() + labs(title = “Box Plot of D_IMF by rs799031002”, x = “rs799031002”, y = “D_IMF”)
p3=ggplot(data, aes(x = rs799031002, y = S_IMF)) + geom_boxplot() + labs(title = “Box Plot of S_IMF by rs799031002”, x = “rs799031002”, y = “S_IMF”)
grid.arrange(p1,p2,p3, ncol=3) # 3개의 그림을 하나의 파일로 통합
# rs209494671와 형질 간의 Box Plot
p1=ggplot(data, aes(x = rs209494671, y = MS)) +
geom_boxplot() +
labs(title = “Box Plot of MS by rs209494671”, x = “rs209494671”, y = “MS”)
p2=ggplot(data, aes(x = rs209494671, y = D_IMF)) +
geom_boxplot() +
labs(title = “Box Plot of D_IMF by rs209494671”, x = “rs209494671”, y = “D_IMF”)
p3=ggplot(data, aes(x = rs209494671, y = S_IMF)) +
geom_boxplot() +
labs(title = “Box Plot of S_IMF by rs209494671”, x = “rs209494671”, y = “S_IMF”)
grid.arrange(p1,p2,p3, ncol=3)
# rs210204777와 형질 간의 Box Plot
p1=ggplot(data, aes(x = rs210204777, y = MS)) +
geom_boxplot() +
labs(title = “Box Plot of MS by rs210204777”, x = “rs210204777”, y = “MS”)
p2=ggplot(data, aes(x = rs210204777, y = D_IMF)) +
geom_boxplot() +
labs(title = “Box Plot of D_IMF by rs210204777”, x = “rs210204777”, y = “D_IMF”)
p3=ggplot(data, aes(x = rs210204777, y = S_IMF)) +
geom_boxplot() +
labs(title = “Box Plot of S_IMF by rs210204777”, x = “rs210204777”, y = “S_IMF”)
grid.arrange(p1,p2,p3, ncol=3)
# rs799031002와 MS 간 분산분석: 본문 내용과 무관하게 표현형과 유전자형(범주형)간의 분산분석 모델
anova_ms <- aov(MS ~ rs799031002, data = data)
summary(anova_ms)
# rs799031002와 D_IMF 간 분산분석
anova_d_imf <- aov(D_IMF ~ rs799031002, data = data)
summary(anova_d_imf)
# rs799031002와 S_IMF 간 분산분석
anova_s_imf <- aov(S_IMF ~ rs799031002, data = data)
summary(anova_s_imf)
# 형질별 요약 통계량 기초통계량을 위한 계산
summary(data$MS)
summary(data$D_IMF)
summary(data$S_IMF)
# SNP별 데이터 분포 요약 유전자형 분포
table(data$rs799031002)
table(data$rs209494671)
table(data$rs210204777)
# 데이터 로드
data <- read.table(“data_snp.txt”, header = TRUE)
# 대립유전자 빈도 계산 함수
calculate_allele_frequency <- function(snp_column) {
# SNP 데이터를 문자로 변환
alleles <- unlist(strsplit(as.character(snp_column), “”))
# 대립유전자별 빈도 계산
freq_table <- table(alleles)
freq_percentage <- prop.table(freq_table) * 100
# 결과 출력
list(allele_counts = freq_table, allele_frequencies = freq_percentage)
}
# rs799031002 대립유전자 빈도 계산(각 유전변이에 대한 빈도계산후 연구자가 직접 표를 작성해야합니다.
rs799031002_freq <- calculate_allele_frequency(data$rs799031002)
cat(“rs799031002 대립유전자 빈도:\n”)
print(rs799031002_freq)
rs209494671_freq <- calculate_allele_frequency(data$rs209494671)
cat(“rs209494671 대립유전자 빈도:\n”)
print(rs209494671_freq)
rs210204777_freq <- calculate_allele_frequency(data$rs210204777)
cat(“rs210204777 대립유전자 빈도:\n”)
print(rs210204777_freq)
# 유전자형 변환 함수: 현재 파일에서는 유전자형이 범주형으로 되어 있어서, 이를 수치형(0,1,2)로 변환하여 회귀분석을 진행해야 해서, 유전자형의 수치형 변환
convert_genotype <- function(genotype_column) {
# 대립유전자 빈도 계산
alleles <- unlist(strsplit(as.character(genotype_column), “”))
allele_freq <- table(alleles)
# Minor allele 확인
minor_allele <- names(allele_freq)[which.min(allele_freq)]
# 유전자형을 숫자로 변환
sapply(genotype_column, function(genotype) {
if (genotype == paste0(minor_allele, minor_allele)) {
return(2) # Minor homozygote
} else if (grepl(minor_allele, genotype)) {
return(1) # Heterozygote
} else {
return(0) # Major homozygote
}
})
}
# 모든 유전자형 열에 변환 적용
genotype_columns <- colnames(data)[5:ncol(data)]
for (col in genotype_columns) {
data[[col]] <- convert_genotype(data[[col]])
}
# 결과 출력
print(data)
# 변환된 수치형자료를 이용하여 회귀분석 수행.
# Regression analysis: rs799031002와 MS 간
R1 <- lm(MS ~ rs799031002, data = data)
summary(R1)
R2 <- lm(D_IMF ~ rs799031002, data = data)
summary(R2)
R3 <- lm(S_IMF ~ rs799031002, data = data)
summary(R3)
R4 <- lm(MS ~ rs209494671, data = data)
summary(R4)
R5 <- lm(D_IMF ~ rs209494671, data = data)
summary(R5)
R6 <- lm(S_IMF ~ rs209494671, data = data)
summary(R6)
R7 <- lm(MS ~ rs210204777, data = data)
summary(R7)
R8 <- lm(D_IMF ~ rs210204777, data = data)
summary(R8)
R9 <- lm(S_IMF ~ rs210204777, data = data)
summary(R9)
등심, 설도는 한우의 물리적인 공간을 나타내는 범주의 이름이며 서로 배타적입니다. 집합으로 설명하면, 부위라는 집합의 등심과 설도는 부분집합입니다. 따라서 부위는 범주형변수의 이름으로, 등심과 설도는 범주형변수의 값(데이터)으로 모델링합니다. 등심과 설도는 부위를 관측한 결과인 범주형데이터입니다.
유전적으로 결정된다면 확률변수로 볼 수 있습니다.
연속형데이터입니다.
범주형데이터입니다